Entwicklung einer netzfreien Galerkinmethode für Zellmechanik mit großen Deformationen

In der Biomechanik werden mechanische Modelle von Zellen, zum Beispiel roten Blutkörperchen verwendet, welche selbst bei wenig komplexen Geometrien mit einer hohen Anzahl finiter Elemente diskretisiert werden müssen (Abbildung 1). Der daraus folgende hohe Rechenaufwand erschwert den Einsatz dieser Modelle für großskalige Simulationen mit vielen Zellen.
Netzfreie Methoden, welche die Geometrie der Zelle mit deutlich weniger nichtlokalen Ansatzfunktionen parametrisieren, können die Anzahl der Freiheitsgrade deutlich reduzieren. Die sogenannten Kugelflächenfunktionen bilden eine Orthonormalbasis von Funktionen auf der Kugeloberfläche, mit denen sich die Oberfläche von im weitesten Sinne kugelartige Gebieten (sog. Sterngebiete) beliebig gut annähern lassen. Oft reichen für eine gute Approximation schon relativ wenige Kugelflächenfunktionen aus (Abbildung 2).
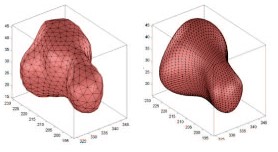
(rechts) [2]

